중국 광둥성 수족구병(HFMD) 역학, 2013-2021
수족구병(HFMD) 보고, 검체 수집 및 분자형 분류 시스템은 그림 1에 나와 있습니다. 주정부 신고 질병 보고 시스템에 따르면, 수족구병(HFMD)에 대해 임상적으로 확인된 총 3,009,233건의 사례가 보고되었습니다. 2013~2021년 중국 광동, 2014년에 가장 많은 사례 보고(N= 429,617)이며 2020년에 가장 낮음(N= 60501)(그림 2a). EV 및 특정 유전자형 EV-A71 및 CVA16에 대한 RT-PCR 테스트는 2013~2016년에 HFMD에서 수집된 의심스러운 샘플에 대해 필요했으며, 이는 2017년에 EV-A71, CVA16 및 CVA6으로 확대되었습니다. HFMD에서 총 36,461개의 의심스러운 샘플이 수집되었습니다. 2013~2021년 기간 동안 프로파일링을 위해 수집된 유전자 검사 중 26,086명(71.54%, 26,086/36,461명)이 HIV 양성인 것으로 밝혀졌습니다. EV 유전자형 분포의 경우 EV-A71과 CVA16의 비율은 시간이 지남에 따라 변동되었습니다. CVA16 전염병은 2014년, 2016년, 2018년, 2021년에 각각 상대적으로 높은 발생률(>25%)로 2년마다 증가하는 것으로 관찰되었습니다(그림 2a). 반면 긍정비율은 감소했다(
중국 광동성 HFMD 사례의 장내 바이러스 분포, 2013-2021. (ㅏ) 2013~2016년 EVA71 및 CVA16 분포, 2017~2021년 EVA71, CVA16 및 CVA6 유전자형 분포; (비) 기타 장내 바이러스 양성(2013~2016년에는 비 EVA71 및 비 CVA16 HFMD, 2017~2021년에는 비 EVA71, 비 CVA16 및 비 CVA6 HFMD) HFMD 사례의 장내 바이러스 유전자형 분포. EV, 장내 바이러스. 이력서, 콕사키바이러스. 수족구병(HFMD).
2008~2013년에 관찰된 수족구병(HFMD)의 병인학적 패턴과는 달리, 2013년부터 EV-A71 및 CVA16 이외의 EV 유전자형 검출이 증가했습니다. 이전 보고서에서는 CVA6의 우세를 강조했습니다. 2013년 말까지 광동성 HFMD(HFMD). [20, 27]. 2013년부터 2021년까지 CVA6 발병률이 지속적으로 증가하는 것은 이 유전자형이 이 지역에서 풍토병이 되고 있음을 시사합니다. 비EV-A71/CVA16/CVA6 EV로 식별된 3,119개 샘플 중 CVA10은 2013년부터 2021년 사이에 가장 큰 비율을 차지했으며, 2018년에 64.60%(500/774)의 보급률로 최고조에 달했습니다. 특히 CVA4는 상당한 상승세를 보이며 2021년 29.82%(243/815)로 가장 높은 검출률을 달성했다. 또한 CVA2, CVA5 및 CVA8은 HFMD가 의심되는 사례에서 흔히 발견되었으며, 이는 중국 광동성에서 HFMD의 다각적인 병인학 패턴을 시사합니다(그림 2B).
2013년부터 2021년까지 CVA10의 확산으로 인해 CVA10 감염의 역학적 특성에 대한 조사가 촉발되었습니다. 2017년부터 2021년까지 지방고시질병감시체계에 신고된 CVA10 감염 사례 614건과 EV-A71 감염 사례 467건을 후향적으로 분석했다. 압도적 다수인 CVA10의 98.37%(604/614)와 EV-A71 감염의 96.36%(450/467)가 5세 미만 어린이에게서 발생했습니다. 특히, CVA10 및 EV-A71 감염에 대한 가장 높은 감염률은 1~2세 그룹에서 관찰되었습니다. CVA10 환자의 평균 연령은 1.8세였으며, 이는 EV-A71 감염 환자의 평균 연령 2.3세보다 상당히 낮습니다.에스P > 0.05), 그림 3과 같습니다.
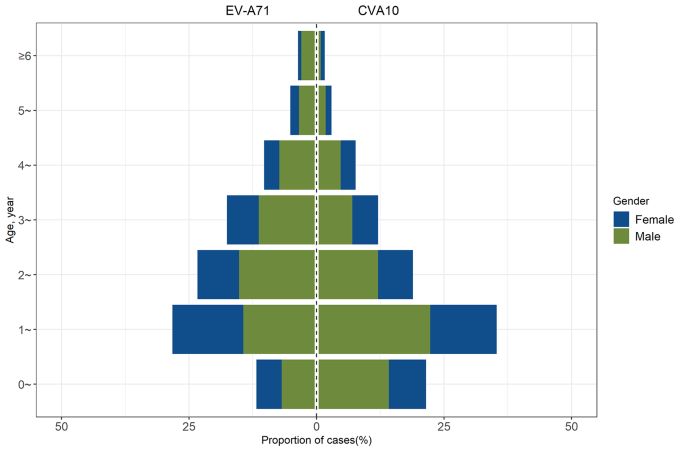
2017년부터 2021년까지 HFMD를 동반한 EVA71 및 CVA10 사례의 연령 및 성별 분포
VP1 유전자와 바이러스 게놈의 계통 발생 구조
현재까지 공개 데이터베이스에는 CVA10에 대한 게놈 서열과 VP1 유전자 서열의 수가 제한되어 있었습니다. 증가하는 전염병과 잠재적으로 관련된 바이러스 및 돌연변이의 진화적 분포에 대한 통찰력을 얻기 위해 우리는 연구에서 2010년부터 2021년까지 수집된 샘플을 나타내는 CVA10 바이러스 게놈의 총 78개의 완전하거나 거의 완전한 서열을 생성했습니다. 분석 공개 데이터베이스에서 이용 가능한 402개의 CVA10 게놈 서열을 포함하여 수행되었습니다.
첫째, 모든 CVA10 게놈 서열은 참조 게놈으로 AY421767에 정렬되었습니다. 이어서 상응하는 VP1 유전자(2477-3377nt)를 스플라이싱하여 계통발생 구조물에 사용했습니다. 우리는 이전 연구의 VP1 분류 체계를 따랐습니다. [28, 29]모든 서열은 6개의 유전자 그룹으로 분류될 수 있습니다. 서로 다른 유전자형 그룹 간의 VP1 유전적 유사성은 74.94~86.47% 범위였으며, 그룹 A와 D 유전자형이 가장 큰 차이를 나타냈습니다(표 1). 모든 광동 CVA10 게놈 서열이 유전자군 C에 속하는 균주에서 풍토병 순환이 관찰되었습니다. 게놈 서열에 따르면 CVA10의 유병률 증가는 2015년 이후 가장 큰 유전자군을 나타내는 유전자군 C 바이러스의 두 하위 그룹에 의해 발생했을 가능성이 가장 높습니다(그림 4a, 상단 패널).
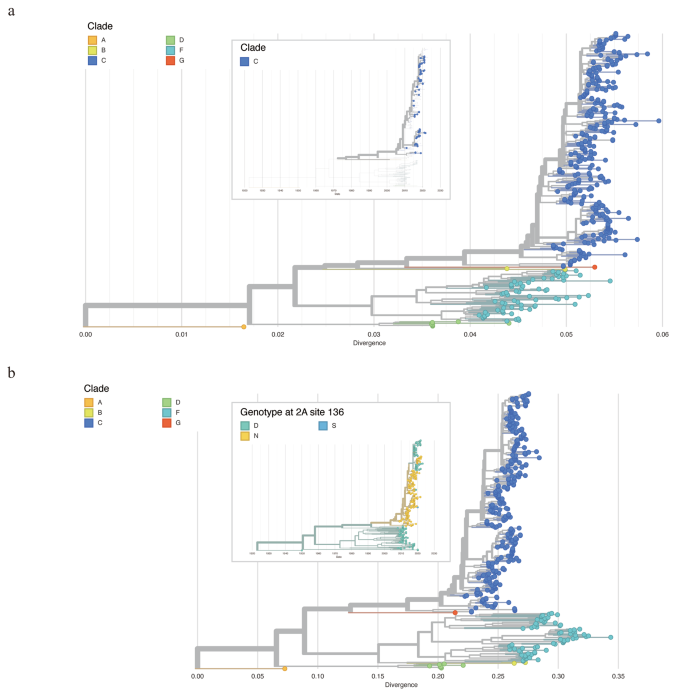
VP1(a) 및 전체 게놈(b)을 기반으로 구축된 CVA10의 최대 우도 트리. 상단 패널의 썸네일(ㅏ)는 VP1의 시간 규모 계통 발생 수를 보여 주며 본 연구에서 생성된 광동 서열만 표시됩니다. 상단 패널의 썸네일(비)는 CVA10 게놈의 진화적 발전을 보여줍니다. 나무는 단백질 2의 136번 위치에 있는 아미노산으로 착색됩니다.
둘째, 우리는 CVA10 게놈 서열을 사용하여 계통 발생을 재구성했으며 VP1 분류 결과에 따라 계통에 주석을 달았습니다 (그림 4b). 최대 가능성 방법을 사용한 조상 서열 재구성은 각 노드에 대한 가능성 있는 조상 서열과 각 가지에서 발생하는 해당 아미노산 돌연변이를 나타냅니다. 흥미롭게도, 조상 서열 재구성은 2017년 이후에 수집된 CVA10 서열의 대부분(202개 중 84개, 41.58%)을 포함하고 독특한 아미노산인 N136D를 포함하는 새로 출현하는 하위 그룹 C를 발생시키는 내부 가지를 밝혀냈습니다. , 단백질 2a에서. 이 비동의 돌연변이는 전체 게놈 서열에서 확인된 104개의 동의 돌연변이와 극명한 대조를 이룹니다. (그림 4b).
특히, 바이러스 진화 과정에서 136번 위치의 아미노산이 반복적으로 변경되었습니다. N136D 돌연변이 및 다른 모든 유전자그룹의 서열을 포함하는 CVA10의 유전자그룹 C로 이어지는 내부 분지는 단백질 2 A의 위치 136에 있는 아스파르트산(Asp, D)이었습니다. 역 돌연변이는 그룹 C As에서 나타나는 새로운 하위그룹에서 관찰될 수 있습니다. 위에 언급했듯이. 또한, 새로 출현하는 코호트의 소규모 그룹이 2A 단백질에 N136D 돌연변이가 있고 3A, 3C 및 3D 단백질에 다른 아미노산 돌연변이가 있음을 관찰했습니다. CVA10 바이러스 진화 메커니즘을 이해하려면 2A 단백질의 위치 136에 있는 돌연변이의 생물학적 기능을 추가로 분석해야 합니다.
CVA10과 다른 장내 바이러스 간의 빈번한 재조합
VP1 서열과 CVA10의 전체 게놈 서열에 대한 유전자 클러스터 주석은 이 두 가지의 진화 구조의 차이를 보여줍니다(그림 4a-b). 상세하게, CVA10 서열의 유전자군 B는 VP1 계통발생에서 유전자군 G 및 유전자군 C와 밀접하게 관련되어 있지만 게놈 서열 서열에서는 유전자군 F 균주와 클러스터링되어 있다. 이러한 불일치는 CVA10 바이러스 진화 동안 유전자 풀 내 재조합 또는 EV 종 간의 재조합 가능성을 강조했습니다. 유전자 재조합에 대한 가능한 단서를 조사하기 위해 우리는 CVA10 게놈의 P1, P2 및 P3 영역을 각각 서열 분석했습니다. 마찬가지로, CVA10의 유전자 클러스터 G는 구조적 단백질 코딩 영역(P1)의 계통발생과 일치하지 않는 비구조적 단백질 코딩 서열(P2 및 P3)의 서열에서 유전자 클러스터 F와 밀접하게 관련되어 있습니다(보충 그림 1a-c). . 비구조적 단백질 코딩 영역의 EV 종들 사이에서 재조합이 흔한 것으로 알려져 있습니다 [30, 31]. CVA10과 다른 EV 유형 간의 관계를 명확히 하기 위해 다른 EV와 밀접하게 관련된 모든 시퀀스를 포함하여 P2와 P3의 시퀀스를 재구성했습니다. P2 영역의 경우 모든 CVA10 유전자 클러스터는 계통 발생 수 전체에 분산되어 있으며 다른 CVA10 유전자 클러스터 시퀀스가 아닌 다른 EV 종과 클러스터되어 종간 재조합 가능성을 강조합니다 (그림 5a). 구체적으로, 유전자군 F의 P2 단편은 3개의 서로 다른 주요 그룹으로 분류되었으며, 이는 CVA10 유전자군 F, CVA2, CVA4, CVA8, EVA114, EVA120 및 기타 유형 간의 재조합 가능성을 나타냅니다. 중국 본토의 주요 CVA10 균주인 Genogroup C는 함께 클러스터되었으며 단 하나의 서열(MF422532)만이 EV-A71 및 CVA2 유형과 재조합될 가능성이 있습니다. F 유전자 클러스터 서열이 다른 EV 유형과 밀접하게 관련되어 있고 여러 그룹으로 클러스터된 P3 영역(그림 5B)에서도 유사한 결과가 발견되었습니다. CVA10의 유전자 클러스터 F와 비구조적 단백질 코딩 서열의 다른 EV 사이의 복잡한 상호작용은 집단 내에서 이 유전자 클러스터의 광범위한 순환 가능성을 나타냅니다. CVA10의 유전자 클러스터 F에서 재조합 이벤트의 존재를 확인하기 위해 다른 EV를 사용하여 Simplot 분석을 수행했습니다(그림 6). 결과는 P2 코딩 영역에서 CVA10, EVA120, CVA2 및 CVA4의 유전자 그룹 F 사이에서 부트스트랩 값이 70%보다 큰 다중 재조합 이벤트를 나타냅니다.
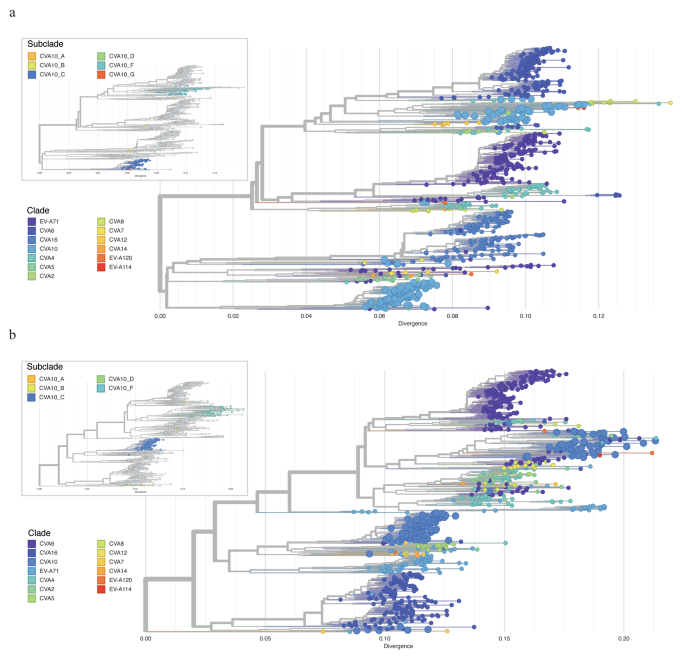
P2(ㅏ) 및 P3(비) CVA10 영역 및 기타 밀접하게 관련된 엔테로바이러스 서열. 마이크로플레이트는 CVA10 하위 계통의 계통발생적 위치를 입증했습니다.

CVA10의 유전자 클러스터 F의 재조합 분석. CVA10의 유전자군 F와 EVA120 사이의 부트스트랩 스캐닝의 재조합 분석(ㅏCVA2(비CVA4(씨). 대표적인 균주(EVA120: MT081367, CVA2: MF422537, CVA4: OM417121)를 각각 쿼리 시퀀스로 사용했습니다. 점선은 70% 부트스트랩 지원을 나타냅니다. CVA10의 게놈 구조는 참조 균주 CVA10 AY421767에 따라 주석을 달았습니다.

“트위터를 통해 다양한 주제에 대한 생각을 나누는 아 동율은 정신적으로 깊이 있습니다. 그는 맥주를 사랑하지만, 때로는 그의 무관심함이 돋보입니다. 그러나 그의 음악에 대한 열정은 누구보다도 진실합니다.”
